Chapitre 7 Modèles linéaires généralisés
7.1 Principe
Les hypothèses du modèle linéaire classique, à savoir normalité et homoscédasticité des résidus, OU une taille d’échantillon suffisammnent grande, ne sont pas toujours vérifiées. C’est notamment le cas pour certains types de variables-réponses comme les variables binaires (oui/non, vrai/faux, 1/0…) ou les variables d’abondance.
Les modèles linéaires généralisés permettent de s’affranchir de ces problèmes pour certains types de variables…
Pour ce faire, ils n’estiment non pas directement les paramètres d’intérêt du modèle (par exemple la moyenne \(\mu\) de la variable réponse), mais plutôt une valeur transformée de ces paramètres, \(f(\mu)\). La fonction qui permet d’opérer cette transformation s’appelle fonction de lien.
La table suivante présente quelques uns des types de variables réponses analysables via un modèle linéaire généralisé.
| Type de réponse | Distribution | fonction R |
| Une variable pouvant prendre seulement 2 valeurs (par exemple 0/1 ou “oui”/“non”) | Bernouilli (binaire) |
glm(y~x, family=binomial(link="logit"))
|
| Une variable pouvant prendre seulement des valeurs entières et positives (0,1,2,3,4,…) | Poisson | glm(y~x, family=binomial(link=“logit”)) |
| Une variable pouvant prendre seulement des valeurs entières et positives (0,1,2,3,4,…) et dont la variance est particulièrement forte | Binomiale négative |
glm.nb(y~x)
|
| Une variable pouvant prendre des valeurs continues et dont la distribution est asymétrique. | Gamma |
glm(y~x, family=Gamma(link = "inverse")))
|
7.2 Exemple 1: distribution binomiale négative
Imaginons que l’on s’intéresse au jeu de données broceliande, et plus particulièrement au lien entre la présence d’un enchantement et le nombre de fées:
ggplot(broceliande, aes(x=enchantement, y=fees))+
geom_boxplot(col="aquamarine")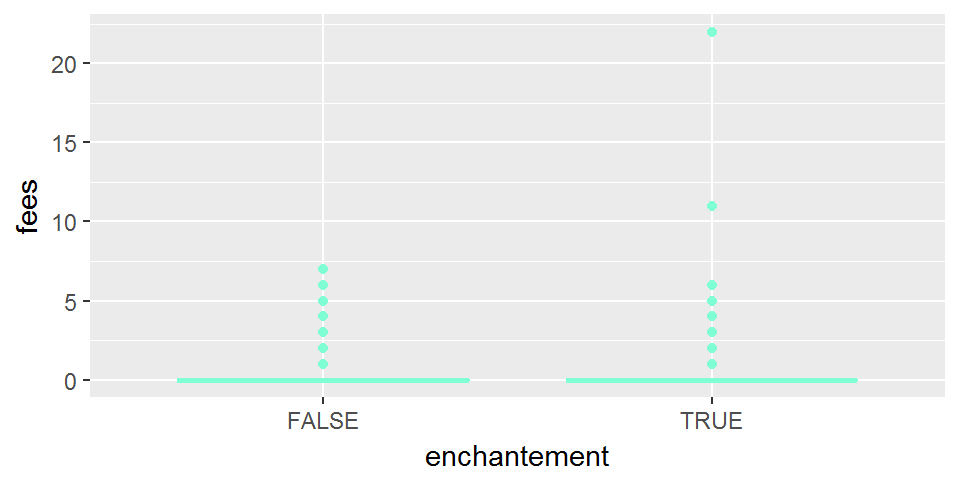
Très clairement, la distribution résiduelle d’un modèle le nombre de fées moyen dépendrait de l’enchantement n’est pas gaussienne… Les fées semblent en outre avoir tendance à se rassembler au sein d’un même arbre (soit par tendance grégaire, soit parce que les arbres en question répondent à des critères de choix qui, peut-être, nous échappent du fait de notre condition de simples mortels).
Et une transformation, type transformation log, n’y changerait rien ! En effet, on a ici affaire à un type de distribution typique des données d’abondance ou de comptage.
Classiquement, le modèle de distribution pour de telles données de comptage peut être soit
- une distribution de Poisson (du nom d’un mathématicien français) si la variance est à peu près égale à la moyenne,
- soit une distribution binomiale négative quand la variance est plus forte que la moyenne.
7.2.1 Représentation
fees_resume=broceliande %>%
group_by(enchantement) %>%
summarise(moy_fees=mean(fees))
ggplot(broceliande, aes(x=fees))+
geom_histogram(fill="aquamarine")+
facet_grid(~enchantement)+
scale_y_sqrt()+
geom_vline(data=fees_resume, aes(xintercept=moy_fees), linetype=3)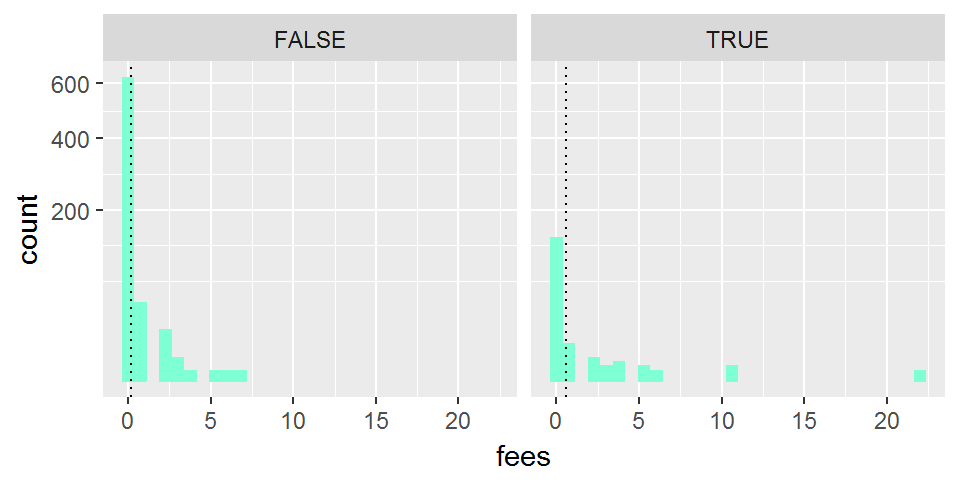
Ici la distribution est très asymétrique et le modèle de distribution le plus adapté semble être une distribution binomiale négative.
7.2.2 Ajustement d’un modèle linéaire généralisé pour une distribution binomiale négative
La fonction glm() de base ne propose pas d’ajuster un modèle avec une telle distribution. On peut utiliser à la place la fonction glm.nb(), dans le package MASS.
library(MASS)
myglm=glm.nb(fees~enchantement+0, data=broceliande)
### estimation de log(mu)
myglm$coeff## enchantementFALSE enchantementTRUE
## -1.8032870 -0.5476396### estimation de mu
exp(myglm$coeff)## enchantementFALSE enchantementTRUE
## 0.1647564 0.5783133On peut vérifier que cette estimation correspond à la moyenne empirique:
fees_resume## # A tibble: 2 x 2
## enchantement moy_fees
## <lgl> <dbl>
## 1 F 0.165
## 2 T 0.578Estimation du paramètre de dispersion inverse (“size”)
myglm$theta## [1] 0.1028071Pour vérifier la significativité du lien entre enchantement et fées:
anova(myglm, test="Chisq")## Analysis of Deviance Table
##
## Model: Negative Binomial(0.1028), link: log
##
## Response: fees
##
## Terms added sequentially (first to last)
##
##
## Df Deviance Resid. Df Resid. Dev Pr(>Chi)
## NULL 864 388.62
## enchantement 2 118.06 862 270.55 < 2.2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Que se serait-il passé si nous avions appliqué un modèle linéaire classique à nos données?
anova(lm(fees~enchantement, data=broceliande), test="Chisq")## Analysis of Variance Table
##
## Response: fees
## Df Sum Sq Mean Sq F value Pr(>F)
## enchantement 1 22.94 22.9361 17.803 2.709e-05 ***
## Residuals 862 1110.53 1.2883
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Eh bien, cela n’aurait pas fondamentalement changé notre conclusion puisque nous aurions là aussi rejeté l’hypothèse nulle (et donc conclu que la présence d’un enchantement a un effet significatif sur le nombre moyen de fées dans un arbre). Cela tend à prouver que nous avions en fait suffisamment de données pour faire abstraction des hypothèses de normalité et homoscedasticité du modèle linéaire…
Si l’on s’intéressait à un jeu de données plus restreint, l’histoire ne serait pas la même (ici, on “restreint” le jeu de données artificillement, en tirant au hasard une centaine d’individus)… Voyez plutôt:
broceliande_restreint=sample_n(broceliande,100)
anova(lm(fees~enchantement,data=broceliande_restreint), test="Chisq")## Analysis of Variance Table
##
## Response: fees
## Df Sum Sq Mean Sq F value Pr(>F)
## enchantement 1 25.316 25.316 15.734 0.0001388 ***
## Residuals 98 157.684 1.609
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1anova(glm.nb(fees~enchantement,data=broceliande_restreint), test="Chisq")## Analysis of Deviance Table
##
## Model: Negative Binomial(0.1618), link: log
##
## Response: fees
##
## Terms added sequentially (first to last)
##
##
## Df Deviance Resid. Df Resid. Dev Pr(>Chi)
## NULL 99 48.413
## enchantement 1 18.076 98 30.337 2.122e-05 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1On voit ici que passer par un modèle linéaire généralisé plutôt que par un modèle linéaire classique n’est pas un luxe de précaution puisque le résultat de test n’est pas le même…
7.3 Exemple 2: distribution binomiale
Imaginons maintenant que l’on souhaite décrire l’existence d’un enchantement en fonction de la poudre de perlimpinpin…
Remarquez que, dans la section 4.1, on expliquait non pas enchantement par perlimpinpin, mais perlimpinpin par enchantement… Ainsi, l’intérêt d’expliquer la variable binaire (enchantement) par la variable quantitative (perlimpinpin) a pour principal intérêt de fournir un moyen de prédire si un arbre est enchanté ou non sur la base de sa concentration en perlimpinpin (il peut être plus facile de doser la poudre de perlimpinpin que de détecter un enchantement pour les simples mortels que nous sommes).
Voyez plutôt:
ggplot(broceliande, aes(x=perlimpinpin,y=enchantement))+
geom_point(col="light blue")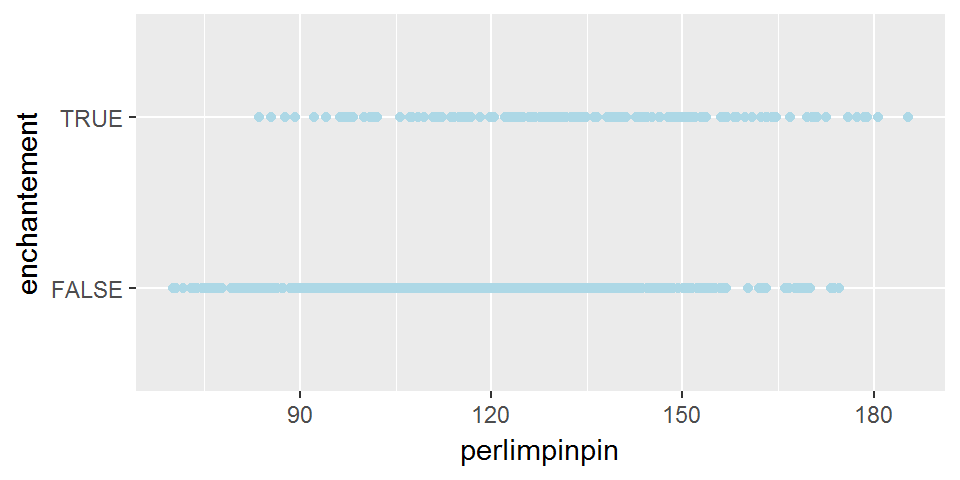
Ici, le graphique semble bien suggérer que plus la concentration en perlimpinpin est forte, et plus les chances que l’arbre soit enchanté est important.
Ainsi, on cherche à décrire la probabilité que l’arbre soit enchanté en fonction de la concentration en perlimpinpin et pour ce faire on passe par une fonction de lien logit.
Ce modèle correspond en fait à ce qu’on appelle une régression logistique.
La fonction logit s’applique en effet à une probabilité (donc quelque chose qui varie entre 0 et 1) et la transforme de la manière suivante:
\[ logit(\pi)=\frac{log(\pi)}{log(1-\pi)} \]
logit=function(pi){
return(log(pi/(1-pi)))
}
inv.logit=function(lpi){
return((exp(lpi))/(1+exp(lpi)))
}Si logit(\(\pi\)) varie linéairement en fonction d’une variable \(X\), alors voilà comment \(\pi\) varie en fonction de \(X\)
datlogit=data.frame(X=seq(-5,5,length.out=1000)) %>%
mutate(logitpi=2*X+3) %>%
mutate(pi=inv.logit(logitpi))
ggplot(datlogit, aes(x=X, y=pi))+geom_line()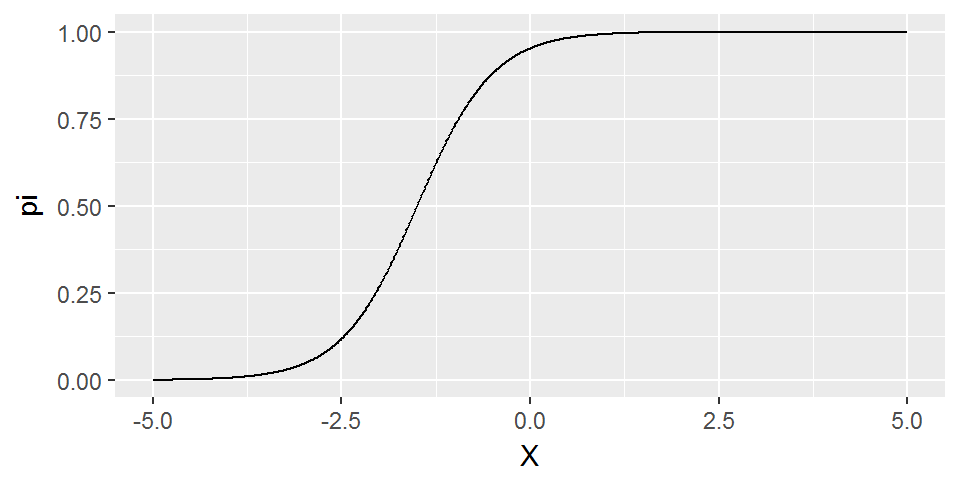
Remarquons que si \(\pi \in\left[0,1\right]\), en revanche, \(logit(\pi)\in\left[-\infty,+\infty\right]\).
Avec un GLM, on va modéliser la valeur de \(logit(\pi)\) :
modele=glm(enchantement~perlimpinpin, data=broceliande, family=binomial(link="logit"))
summary(modele)##
## Call:
## glm(formula = enchantement ~ perlimpinpin, family = binomial(link = "logit"),
## data = broceliande)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -1.3289 -0.6793 -0.5115 -0.3361 2.4434
##
## Coefficients:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) -5.945575 0.580539 -10.241 < 2e-16 ***
## perlimpinpin 0.036045 0.004436 8.126 4.46e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for binomial family taken to be 1)
##
## Null deviance: 845.50 on 863 degrees of freedom
## Residual deviance: 770.71 on 862 degrees of freedom
## AIC: 774.71
##
## Number of Fisher Scoring iterations: 4On retrouve bien l’existence d’un lien significatif entre perlimpinpin sur enchantement.
Par ailleurs, les coefficients renvoyés correspondent aux valeurs estimées de \(logit(\pi)\). Ces valeurs sont modélisées comme dépendant linéairement de perlimpinpin (cf graphique de gauche). Pour obtenir non plus les valeurs estimées de \(logit(\pi)\) mais les valeurs de \(\pi\) elles-mêmes, on peut utiliser la fonction inv.logit (graphique de droite):
ggplot(mutate(broceliande, enchantement=as.numeric(enchantement)),
aes(x=perlimpinpin, y=enchantement)) +
geom_point(col="light blue")+
geom_smooth(method="glm",method.args=list(family="binomial"))
Ce modèle permet ainsi d’estimer que pour avoir une chance sur 2 de tomber sur un arbre enchanté, alors il faut que la quantité de perlimpinpin soit > 165\(\mu\)g/L…