
Le test de Scheffé est un test qu'on applique souvent après une ANOVA: on parle de test post-hoc (au même titre, par exemple, qu'un test de Tukey). En effet, l'ANOVA à 1 facteur permet de mettre en évidence (le cas échéant) le fait qu'au moins un groupe a une moyenne différente des autres. Si on a affaire à 3 groupes ou plus, une question se pose alors: quels sont les groupes différents deux à deux?
Le test de Scheffé, en pratique:
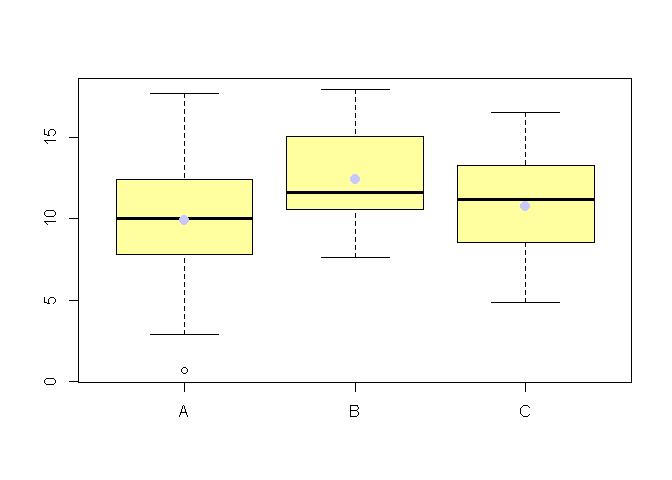
Testons la significativité de l'effet de X sur Y à travers une ANOVA:
mymod=lm(Y~X)
anova(mymod)
## Analysis of Variance Table
##
## Response: Y
## Df Sum Sq Mean Sq F value Pr(>F)
## X 2 61 30.7 2.55 0.087 .
## Residuals 59 710 12.0
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
On rejette l'hypothèse d'égalité de toutes les moyennes au seuil de α = 10%.
Il n'existe pas à l'heure actuelle de test de Scheffé sous R.
(en fait, si, il y a une fonction scheffe.test dans un package qui s'appelle agricolae, mais il me semble après l'avoir testée qu'il y ait une petite erreur dans cette fonction, alors en attendant que son auteur la corrige -ça ne devrait pas être bien long- on va faire comme s'il n'y avait rien...).
Je vous propose d'utiliser une petite fonction que j'ai écrite (vous trouverez des explications sur le code dans le paragraphe suivant) et disponible ici.
Voici comment utiliser cette fonction:
source("test_Scheffe_maison.R")
Scheffe.test(X,Y,alpha=0.1)
## contrast sigma LCI UCI pval
## A-B -2.555 1.133 -5.034 -0.076 0.08718
## A-C -0.868 1.014 -3.087 1.352 0.69507
## B-C 1.688 1.161 -0.854 4.229 0.35448
Les résultats du test de Scheffé montrent que la différence de moyenne entre A et B est significative au seuil de α = 10%. En revanche on n'est pas en mesure d'affirmer que B est différent de C ou que A est différent de C.
Le test de Scheffé, en théorie
Maintenant que vous avez vu comment (et pourquoi) effectuer un test de Scheffé, revenons "en arrière" pour approfondir le principe de ce test.
On considère r groupes, et l'on définit μi comme étant la moyenne du groupe numéro i. Un contraste se définit de la manière suivante:
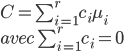
Cela signifie que si l'on a trois groupes, on peut considérer (entre autres) les contrastes suivants:
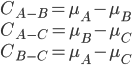
La valeur estimée d'un contraste est simplement:
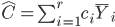
Donc les estimations des trois contrastes qui nous intéressent sont:
estimated_mu=as.vector(tapply(Y,X,"mean"))
contrasts=c(estimated_mu[1]-estimated_mu[2],
estimated_mu[1]-estimated_mu[3],
estimated_mu[2]-estimated_mu[3])
print(contrasts)
## [1] -2.5551 -0.8675 1.6876
La variance associée à cet estimateur est:
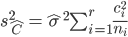
Donc l'écart-type associée à chacun des trois estimateurs considérés est:
n=as.vector(tapply(Y,X,"length"))
N=sum(n)
r=3
estimated_sigma=sqrt(sum(mymod$residuals^2)/(N-r))
sigma_contrasts=c(sqrt(estimated_sigma^2*(1/n[1]+1/n[2])),
sqrt(estimated_sigma^2*(1/n[1]+1/n[3])),
sqrt(estimated_sigma^2*(1/n[2]+1/n[3])))
print(sigma_contrasts)
## [1] 1.133 1.014 1.161
Ainsi il y a une probabilité de 1 − α que C se trouve dans l'intervalle suivant:
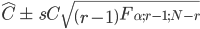
On peut donc calculer les intervalles de confiance comme suit:
alpha=0.1
delta=sigma_contrasts*sqrt(2*qf(1-alpha,3-1,length(X)-3))
results=data.frame(round(contrasts,3),
round(sigma_contrasts,3),
round(contrasts-delta,3),
round(contrasts+delta,3))
row.names(results)=c("A-B","A-C","B-C")
colnames(results)=c("contrast","sigma","LCI","UCI")
Par ailleurs la statistique suivante:
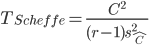
suit une loi de Fisher sous hypothèse que tous les contrastes sont nuls.
On peut donc calculer les p-values comme suit:
T_Scheffe=((contrasts^2)/(sigma_contrasts^2))/(3-1)
pvalues=1-pf(T_Scheffe, 3-1,length(X)-3)
print(data.frame(results,pvalues))
## contrast sigma LCI UCI pvalues
## A-B -2.555 1.133 -5.034 -0.076 0.08718
## A-C -0.868 1.014 -3.087 1.352 0.69507
## B-C 1.688 1.161 -0.854 4.229 0.35448
1 Comment
Andréa
Bonjour,
Merci pour cet article. Il semble que le test en question existe sur R finalement (package DescTool)s