Thomas Gibaud
Abstract: Nature excels at building complex functional materials. We however are far from understanding
the structure and mechanism involved in those materials and even further from being able to replicate equivalent materials.
Here I will present two bottom up approaches that shed light into complex biological structures and mechanisms.
Both those approach will rely on colloidal science. I will therefore progressively introduce model colloidal systems,
the theoretical and experimental tools to study them while explicitly illustrating the two approaches with examples.
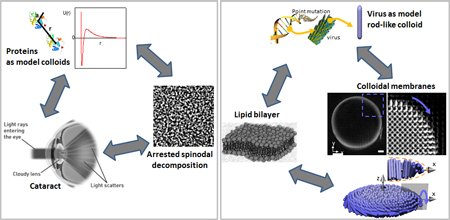
The first approach consist in purifying and isolating the constitutive molecules of the biological structures,
to characterize the individual properties of each molecules and to find the minimalistic number of different molecules
that are necessary to reproduce some properties of the biological assemblage.
We will discuss the case of cataract, a disease that causes blindness. In the second approach,
we will use colloids as minimalist building blocks to mimic biological structures.
The colloidal nature of the building blocks allows up-scaling the size and slowing the dynamics of the resulting
self-assembled structure allowing better visualization, manipulation and modelization.
We will discuss the case of lipid membranes, the main component of the cells wall.
Data analysis:
- perrin.tif - This sequence of images is from the historical experiments of Jean Perrin. Extract the barometric law and the Avogadro number.
Caractérisation des propriétés dynamiques et mécaniques de quelques fluides: de l’eau, des micelles géantes et du yaourt: Dans ces travaux pratiques nous vous proposons de caractériser la dynamique de traceurs colloïdaux par suivie de leur trajectoire au cours du temps dans différents fluides viscoélastiques. A travers ces mesures nous en déduirons les propriétés mécaniques des fluides viscoélastiques : viscosité et module élastique qui seront comparées à des mesures plus classique effectué à l’aide d’un rhéomètre. Ces techniques sont couramment utilisées en matière molle et en biologie.
Ces mesures seront appliquées à trois types de fluide : pour commencer, l’eau un fluide newtonien ; puis une solution de micelle géante, un fluide viscoélastique parfaitement modélisé par le model de Maxwell ; et finalement du yaourt comme un exemple de fluide qui gélifie au cours du temps.
Mot clefs : colloïdes, microscopie, rhéologie, suivie de particules, mouvement brownien, viscosité, module élastique
Caractérisation des propriétés dynamiques d’une solution de bactéries: Dans ces travaux pratiques nous proposons de mesurer quelques propriétés dynamiques d’une solution de bactéries (E. colis) par « Differential Dynamic Microscopy » (DDM). La DDM est une nouvelle technique d’analyse d’images de microscopie qui permet ‘facilement’ de caractériser la dynamique d’un grand nombre de particules. Dans un premier temps on mettra en place cette technique de microscopie et on la testera sur une dispersion colloïdale, des particules passives qui suivent un mouvement Brownien. On mesurera le coefficient de diffusion de ces colloïdes et la viscosité du solvant. Une fois la technique validé on mesurera les propriétés d’une suspension de particules actives, des bactéries. En particulier, on mettra en évidence le régime balistique au temps cours, lié à la propulsion rectiligne des bactéries avec leurs flagelles, ainsi que le régime diffusif lié à la désynchronisation sporadique des flagelles.
Mot clefs : colloïdes, microscopie, mouvement brownien, viscosité, 'differential dynamic microscopy', bactéries