package braidHymo (fr)
braidHymo_fr.RmdFormat des données
Un (1 x rivdata) ou deux types de données (n x rivdata + rivers) sont utilisés par ce package:
Fichiers rivdata
Il y a autant de fichiers de type rivdata que de rivières considérées dans l’analyse.
Un fichier rivdata correspond à:
- 1 ligne par point d’échantillonnage avec mesure d’altitude
-
plusieurs colonnes, parmi lesquelles 2 sont non-optionnelles et devraient correspondre à:
- un identifiant de section transverse pour chaque point d’échantillonnage (nommé, par défaut,
ID) - une mesure d’altitude pour ce point (nommé, par défaut,
RASTERVALU)
- un identifiant de section transverse pour chaque point d’échantillonnage (nommé, par défaut,
Fichier rivers (optionnel)
Pour calculer les indices sur plusieurs rivières, il est souhaitable de fournir une table rivers contenant diverses informations à propos de ces rivières.
Cette table devrait comprendre:
-
n lignes (autant qu’il y a de fichiers
rivdata) -
plusieurs colonnes, parmi lesquelles 3 sont non-optionnelles et devraient correspondre à:
- la taille du bassin versant en km² (nommée, par défaut,
area) - l’espace séparant les points sur les transects en m (nommé, par défaut,
points_space) - le chemin vers le fichier
rivdata(nommé, par défaut,filepath)
- la taille du bassin versant en km² (nommée, par défaut,
Les autres colonnes peuvent faire référence par exemple au nom de la rivière, à l’année d’échantillonnage, etc.
rivers=readr::read_csv("../data-raw/rivers.csv") %>%
mutate(filepath=paste0("../data-raw/",filepath)) %>%
mutate(year=as.factor(year))
#> Rows: 2 Columns: 6
#> ── Column specification ────────────────────────────────────────────────────────
#> Delimiter: ","
#> chr (3): river, reach, filepath
#> dbl (3): year, area, points_space
#>
#> ℹ Use `spec()` to retrieve the full column specification for this data.
#> ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
# Montre le tableau resultant:
rivers
#> # A tibble: 2 × 6
#> river reach year area points_space filepath
#> <chr> <chr> <fct> <dbl> <dbl> <chr>
#> 1 Drac Chabottes 2018 253 1 ../data-raw/Drac_Chabottes_2018.txt
#> 2 Durance Brillanne 2017 7850 1 ../data-raw/Durance_Brillanne_2017…Une seule rivière: utiliser braidHymo_one()
En premier lieu, lire et nettoyer les données rivdata avec braidHymo_read()
data_Drac=braidHymo_read("../data-raw/Drac_Chabottes_2018.txt")
# Montre les premières lignes de la table:
head(data_Drac)
#> # A tibble: 6 × 2
#> ID_XS Z
#> <int> <dbl>
#> 1 232 1030.
#> 2 231 1030.
#> 3 231 1030.
#> 4 231 1030.
#> 5 231 1030.
#> 6 231 1030.Puis calculer les indices pour cette rivière avec braidHymo_one():
result_Drac=braidHymo_one(data_Drac,
area=253,
points_space=1)
#> Warning in braidHymo_one(data_Drac, area = 253, points_space = 1): The cross-
#> sections with ID 232 contain only one measure.
# Montre les premières lignes de la table:
head(result_Drac)
#> # A tibble: 6 × 6
#> Nb_mean_meas XS_onlyone variable type stat value
#> <dbl> <dbl> <chr> <chr> <chr> <dbl>
#> 1 109. 0.431 BRI_mean BRI* mean 0.00347
#> 2 109. 0.431 BRI_SD BRI* SD 0.00187
#> 3 109. 0.431 BRI_min BRI* min 0.00114
#> 4 109. 0.431 BRI_max BRI* max 0.0136
#> 5 109. 0.431 W_mean W* mean 9.50
#> 6 109. 0.431 W_SD W* SD 2.71Plusieurs rivières: utiliser braidHymo()
Ici, on généralise le processus à 2 rivières.
La fonction braidHymo() récupère toute les informations nécessaires à propos des rivières (et notamment le chemin vers les fichiers rivdata) depuis la table rivers
result=braidHymo(rivers)
#> Warning in .f(rivdata = .l[[1L]][[i]], area = .l[[2L]][[i]], points_space
#> = .l[[3L]][[i]], : The cross-sections with ID 232 contain only one measure.
# Montre les premières lignes de la table
head(result)
#> # A tibble: 6 × 12
#> river reach year area points_space filepath Nb_mean_meas XS_onlyone variable
#> <chr> <chr> <fct> <dbl> <dbl> <chr> <dbl> <dbl> <chr>
#> 1 Drac Chab… 2018 253 1 ../data… 109. 0.431 BRI_mean
#> 2 Drac Chab… 2018 253 1 ../data… 109. 0.431 BRI_SD
#> 3 Drac Chab… 2018 253 1 ../data… 109. 0.431 BRI_min
#> 4 Drac Chab… 2018 253 1 ../data… 109. 0.431 BRI_max
#> 5 Drac Chab… 2018 253 1 ../data… 109. 0.431 W_mean
#> 6 Drac Chab… 2018 253 1 ../data… 109. 0.431 W_SD
#> # … with 3 more variables: type <chr>, stat <chr>, value <dbl>On peut alors comparer graphiquement les résultats des différentes rivières en utilisant la fonction braidHymo_plot():
braidHymo_plot(result,index="BRI*", position=year, color=river)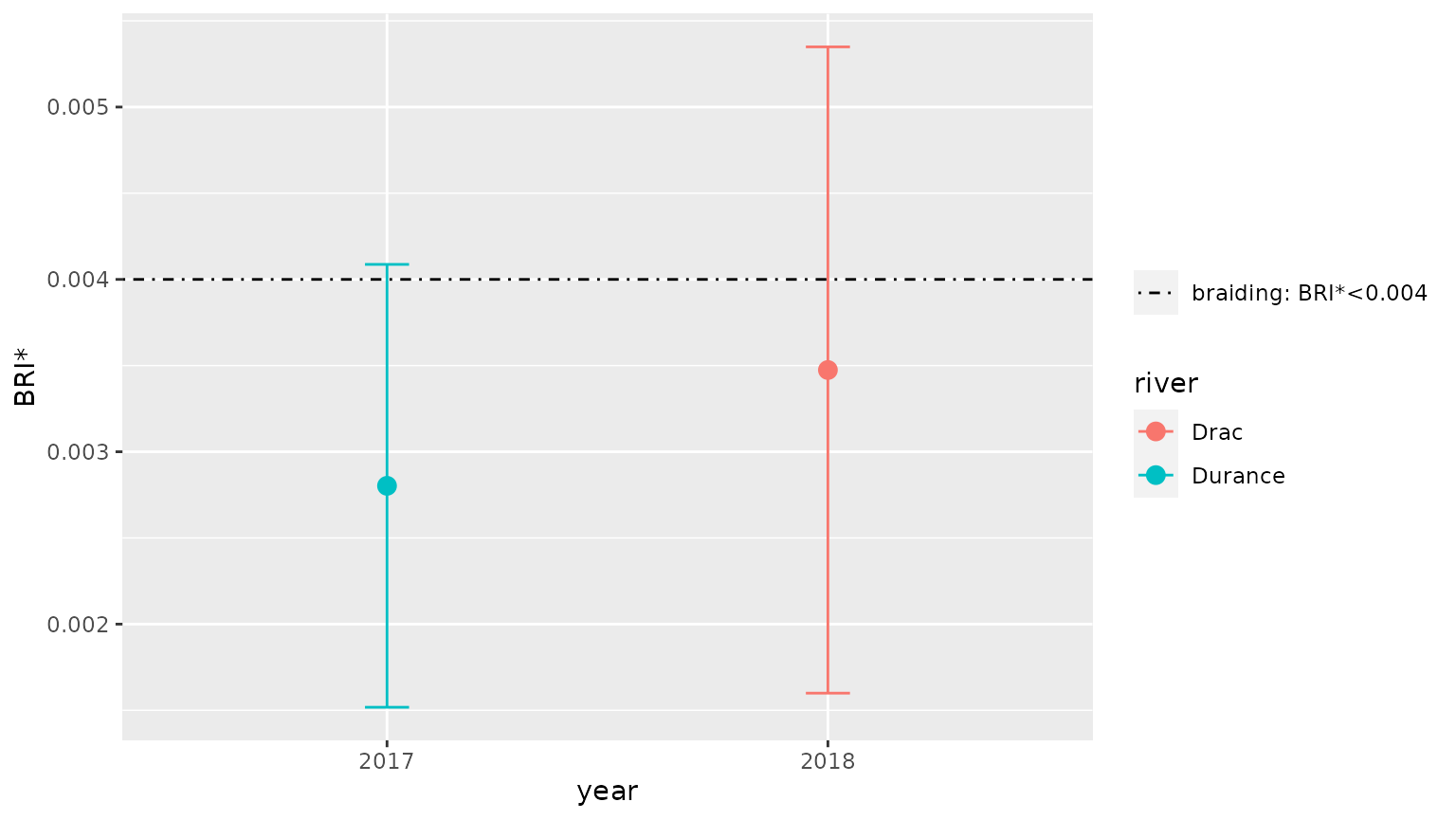
braidHymo_plot(result,index="W*", position=year, color=river)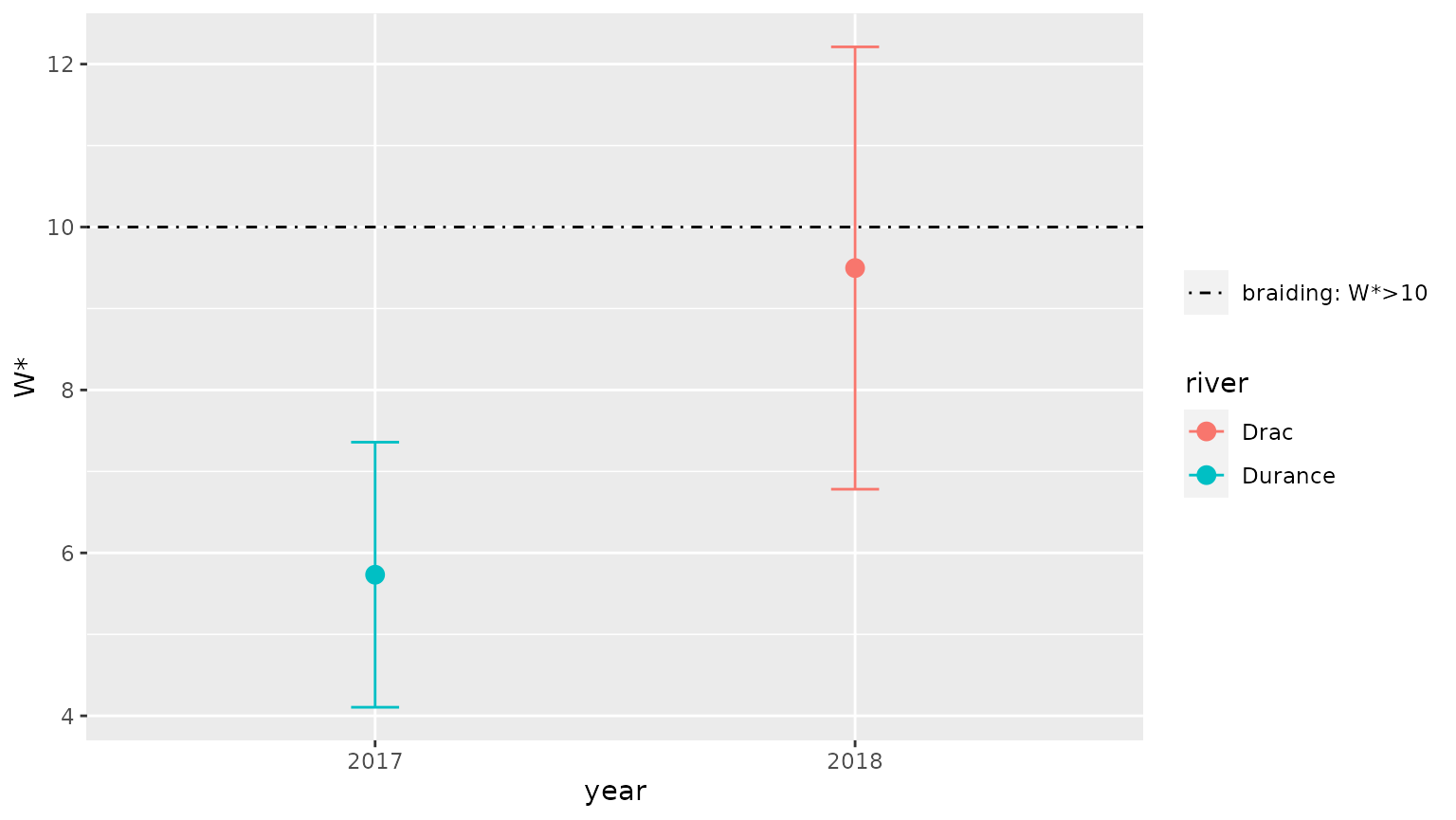
References
When using this package, please refer to Devreux et al. (2021).