Chapitre 6 Modéliser le lien entre plusieurs variables explicatives et une variable réponse
Si les modèles multifactoriels reposent en grande partie sur des principes similaires à ceux des modèles “simples”, ils sont légèrement plus difficiles à appréhender car il est difficile de produire des représentations graphiques de ces modèles. En effet, s’il est simple de représenter \(Y\) en fonction de \(X\), il devient délicat de représenter \(Y\) en fonction de \(X_1\) et \(X_2\) (et, a fortiori, en fonction de \(X_1\),\(X_2\),\(X_3\), etc.).
Néanmoins, moyennant quelques efforts, et en utilisant astucieusement les possibilités graphiques de R, il est possible de visualiser un certain nombre d’éléments en lien avec ces modèles. Nous allons voir des exemples de tels graphiques dans les paragraphes suivants.
Une autre difficulté inhérente aux modèles multifactoriels est liée aux possibles interactions entre variables explicatives.
6.1 Plusieurs variables quantitatives comme variables explicatives: régression multiple

|
Intéressons-nous au jeu de données p_alteration (c’est-à-dire que la variable réponse est quantitative) et des quantités d’ingrédients i_givreboises, i_bave_crapaud, i larmes_crocodile, etc. (c’est-à-dire que les variables explicatives sont quantitatives également), on entre dans le cadre d’une régression linéaire multiple:
|
Je commence par arranger le jeu de données potions pour ne garder que les ingrédients (je log-transforme les quantités d’ingrédients car leur distribution est particulièrement asymétrique) et la propriété p_alteration. J’obtiens le jeu de données ingredients_alteration
potions_i=potions %>%
select(starts_with("i")) %>%
mutate_all("log")
ingredients_alteration=bind_cols(select(potions,p_alteration),
potions_i)
head(ingredients_alteration)## p_alteration i_bave_crapaud i_sang_hirondelle i_aile_papillon
## 1 4.330326 1.943401 2.404449 1.3456830
## 2 4.980520 2.569600 2.778596 -1.3040610
## 3 8.276635 2.747928 2.894108 3.0506434
## 4 4.486441 2.545849 3.084747 3.4421712
## 5 4.041440 1.940331 2.143168 2.4091744
## 6 6.464358 3.069285 3.155645 0.6887613
## i_ectoplasme i_graisse_troll i_pied_lutin i_givreboises i_pierre_lune
## 1 0.78696026 1.683154 2.6924992 3.606648 11.967086
## 2 1.22088567 1.073625 3.1351813 4.599541 15.670782
## 3 0.56914148 2.810524 7.8345469 3.430517 5.962957
## 4 -0.06769568 2.624523 1.5772473 6.294926 23.655917
## 5 2.10065386 1.936514 3.4060533 2.262423 44.477702
## 6 -1.35852116 2.692627 0.3225122 1.309043 58.735741
## i_larmes_crocodile
## 1 -1.1633517
## 2 1.4800278
## 3 0.7567693
## 4 -0.5346780
## 5 3.0079470
## 6 1.97001936.1.1 Représentation graphique
6.1.1.1 Représentation des nuages de points variable explicative vs réponse
ingr_alt_var_as_mod=ingredients_alteration %>%
gather("ingredient","x",starts_with("i_"))
ggplot(ingr_alt_var_as_mod, aes(x=x, y=p_alteration, col=ingredient))+
geom_point()+
geom_smooth(method="lm")+
facet_wrap(~ingredient,scales="free")+
theme(legend.position="none")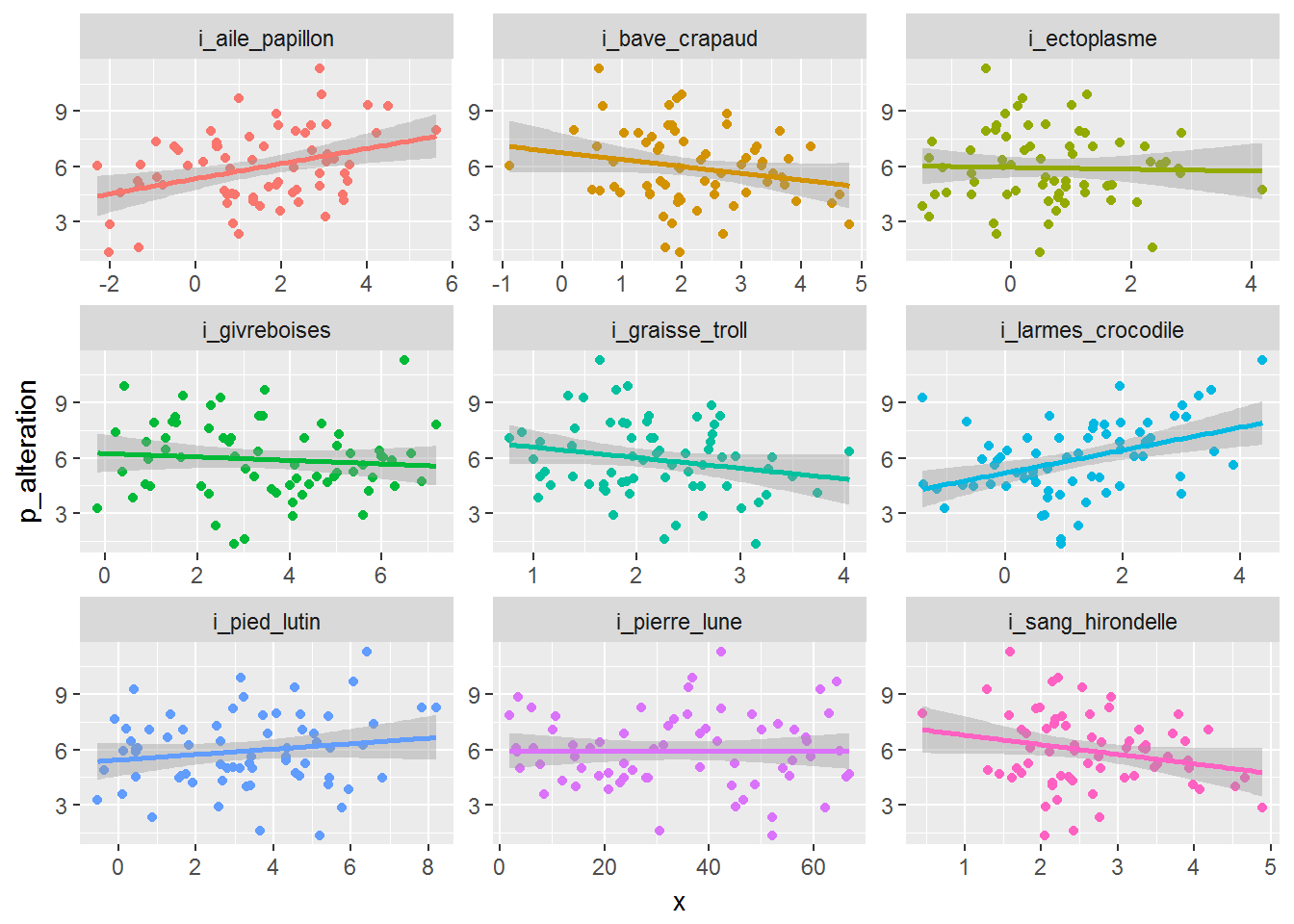
6.1.1.2 Représentation des corrélations
library(corrplot)
res=cor(ingredients_alteration)
corrplot(res,
type = "upper",
diag=FALSE,
tl.col = "dark grey", tl.srt = 45)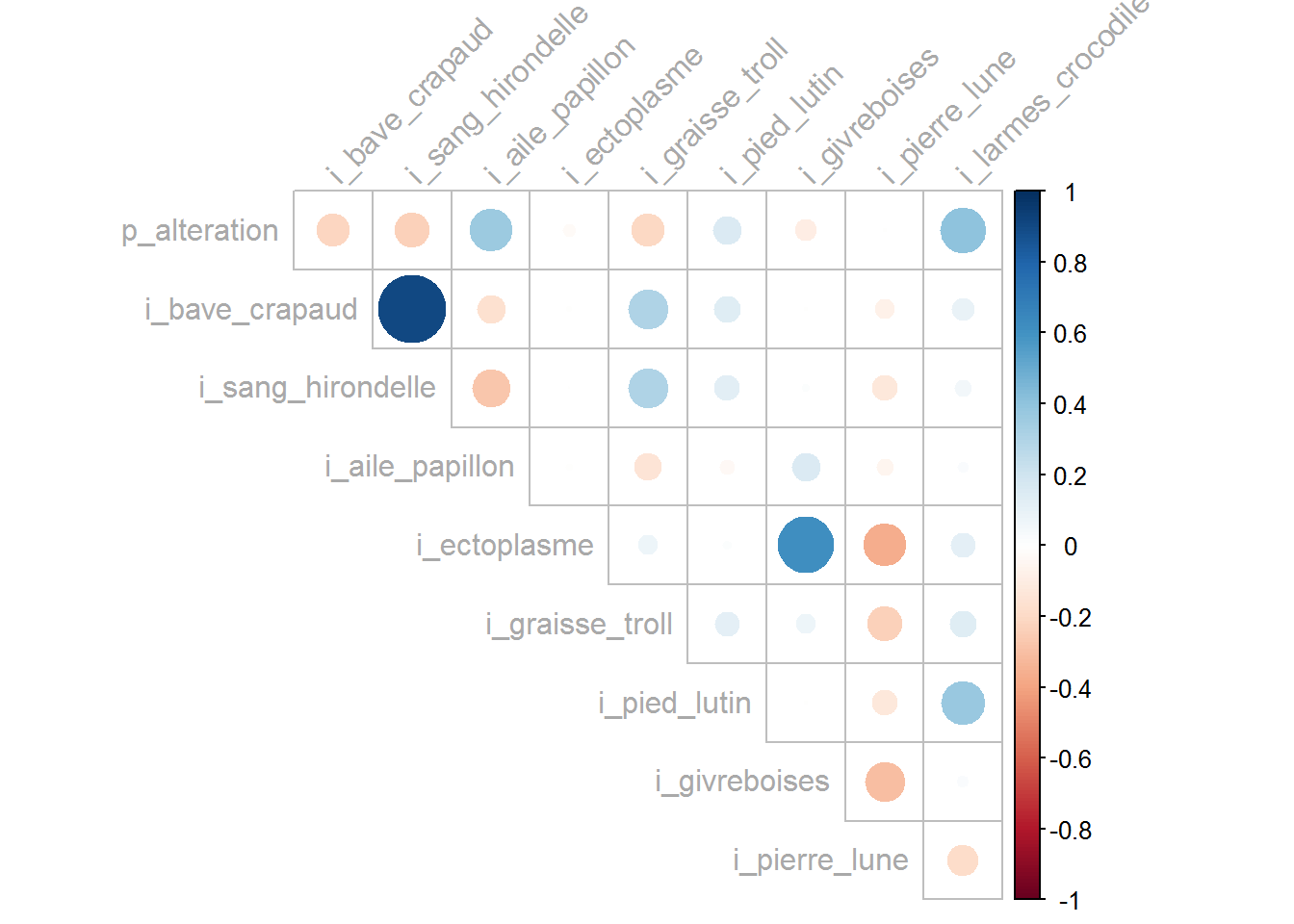
6.1.2 Réalisation de la régression multiple
Pour réaliser une régression multiple sur R, on utilise (encore une fois) la fonction lm()
On adapte simplement la formule pour prendre en compte plusieurs variables explicatives de la manière suivante:
myregmult=lm(p_alteration~i_bave_crapaud+i_pierre_lune+i_givreboises,
data=ingredients_alteration)Si l’on veut considérer un grand nombre de variables explicatives, il peut être plus aisé de passer par la syntaxe suivante (avec laquelle l’ensemble des variables de ingredients_alteration -à l’exception de la variable réponse bien sûr-, est utilisé):
myregmult=lm(p_alteration~.,data=ingredients_alteration)
summary(myregmult)##
## Call:
## lm(formula = p_alteration ~ ., data = ingredients_alteration)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.8938 -0.9511 0.2420 1.0204 3.1065
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 6.087122 1.341244 4.538 2.97e-05 ***
## i_bave_crapaud -0.522925 0.451946 -1.157 0.252078
## i_sang_hirondelle 0.356494 0.585543 0.609 0.545059
## i_aile_papillon 0.400812 0.130937 3.061 0.003360 **
## i_ectoplasme 0.078413 0.228044 0.344 0.732224
## i_graisse_troll -0.453182 0.312326 -1.451 0.152265
## i_pied_lutin 0.050759 0.105307 0.482 0.631641
## i_givreboises -0.174196 0.141438 -1.232 0.223152
## i_pierre_lune 0.002885 0.012206 0.236 0.814030
## i_larmes_crocodile 0.644605 0.173501 3.715 0.000464 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.699 on 57 degrees of freedom
## Multiple R-squared: 0.3846, Adjusted R-squared: 0.2874
## F-statistic: 3.958 on 9 and 57 DF, p-value: 0.0005823D’après ce modèle, la présence d’ailes de papillon et de larmes de crocodile a un effet significatif sur la propriété d’altération de la potion.
Pourtant, à la vue du graphique 6.1.1.1, on pouvait s’attendre à avoir d’autre ingrédients avec un effet significatif sur cette propriété alchimique… Et, de fait, si l’on fait des modèles de régression linéaire simple pour chaque ingrédient, alors il semble qu’en réalité d’autres ingrédients que ces deux là aient un effet significatif.
nested_ingr=ingr_alt_var_as_mod %>%
group_by(ingredient) %>%
nest()
pval=nested_ingr %>%
pull(data) %>%
map(~summary(lm(p_alteration~x,data=.))$coeff[2,4]) %>%
unlist()
bind_cols(ingredient=nested_ingr %>% pull(ingredient) %>% unlist(),
pval=pval)## # A tibble: 9 x 2
## ingredient pval
## <chr> <dbl>
## 1 i_bave_crapaud 0.0862
## 2 i_sang_hirondelle 0.0574
## 3 i_aile_papillon 0.00264
## 4 i_ectoplasme 0.812
## 5 i_graisse_troll 0.0900
## 6 i_pied_lutin 0.200
## 7 i_givreboises 0.467
## 8 i_pierre_lune 0.983
## 9 i_larmes_crocodile 0.0005976.1.3 Interaction entre les variables explicatives
Observez au passage que les deux lignes de commande suivantes produisent exactement le même résultat (qui n’est pas dépendant de l’ordre dans lequel on introduit les variables explicatives).
summary(lm(p_alteration ~ i_aile_papillon+i_larmes_crocodile), data=ingredients_alteration)
summary(lm(p_alteration ~ i_larmes_crocodile+i_aile_papillon), data=ingredients_alteration)Il est en revanche (comme on l’a déjà vu) dépendant du nombre de variables explicatives introduites.
On a ici un problème dit de colinéarité car nos variables explicatives ne sont pas seulement corrélées à la réponse, mais sont également corrélées entre elles.
6.2 Plusieurs variables explicatives, toutes catégorielles: ANOVA à plusieurs facteurs
On s’intéresse ici au jeu de données brocéliande et on va tenter d’expliquer la quantité de poudre de perlimpinpin en fonction de l’enchantement de l’arbre (vrai ou faux) et de son espèce (chêne, hêtre, sapin, châtaignier). On va donc réaliser une ANOVA à deux facteurs.
6.2.1 Représentation
Pour un nombre limité de variables explicatives (par exemple 2), il est encore possible de représenter graphiquement le modèle en “rusant” à l’aide des fonctionnalités de ggplot2:
broc_summary=broceliande %>%
group_by(enchantement, espece) %>%
summarise(moy_perlimpinpin=mean(perlimpinpin))
ggplot(broceliande, aes(x=enchantement, y=perlimpinpin, fill=espece))+
geom_boxplot()+
geom_point(data=broc_summary, aes(y=moy_perlimpinpin),
position=position_dodge(width=0.75),col="grey",size=2)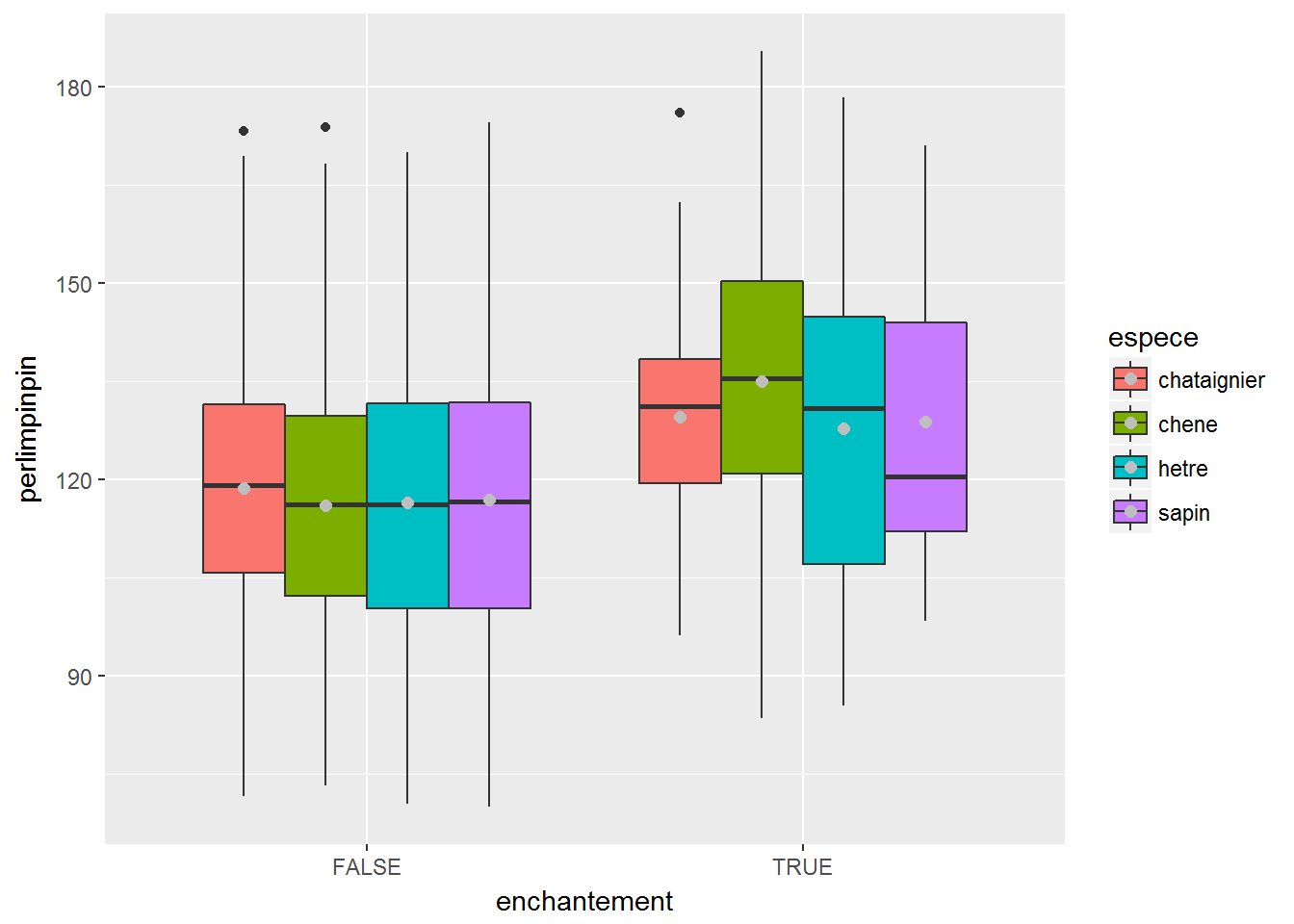
6.2.2 Réalisation de l’ANOVA à deux facteurs
mylm=lm(perlimpinpin~enchantement+espece,data=broceliande)
summary(mylm)##
## Call:
## lm(formula = perlimpinpin ~ enchantement + espece, data = broceliande)
##
## Residuals:
## Min 1Q Median 3Q Max
## -49.561 -14.532 -0.191 14.412 58.134
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 118.191 1.294 91.325 <2e-16 ***
## enchantementTRUE 16.091 1.879 8.563 <2e-16 ***
## especechene -1.149 1.746 -0.658 0.511
## especehetre -2.099 2.032 -1.033 0.302
## especesapin -1.690 2.737 -0.617 0.537
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 20.82 on 859 degrees of freedom
## Multiple R-squared: 0.08611, Adjusted R-squared: 0.08186
## F-statistic: 20.23 on 4 and 859 DF, p-value: 6.071e-16anova(mylm, test="chisq")## Analysis of Variance Table
##
## Response: perlimpinpin
## Df Sum Sq Mean Sq F value Pr(>F)
## enchantement 1 34562 34562 79.7464 <2e-16 ***
## espece 3 517 172 0.3978 0.7546
## Residuals 859 372294 433
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Les tableaux renvoyés sont, grosso modo, de la même forme que ceux renvoyés par l’anova à un facteur, si ce n’est qu’au lieu de comparer seulement 2 modèles, on compare 3 modèles emboîtés 2 à 2. Les résultats correspondent ainsi à:
- la comparaison des modèles 0 et 1 (1ère ligne du tableau),
- la comparaison des modèles 1 et 2 (2ème ligne du tableau),
- la description du modèle complet, i.e. le modèle 2, à travers la somme des résidus (3ème ligne du tableau)
où les modèles en question sont dits “emboîtés 2 à 2”:
- {modèle 0}={perlimpinpin \(\sim\) 1}
- {modèle 1}={perlimpinpin \(\sim\) enchantement}
- {modèle 2}={perlimpinpin \(\sim\) enchantement + espece}
| Variable | ddl | SCsurddl | Fvalue | pvalue | |
|---|---|---|---|---|---|
| 1 | Ajout de la variable \(X_1\) | \(ddl_0-ddl_1\) | \(\frac{SC_0-SC_1}{ddl_0-ddl_1}\) | \(F_{0-1}\) | \(pval_{0-1}\) |
| 2 | Ajout de la variable \(X_2\) | \(ddl_1-ddl_2\) | \(\frac{SC_1-SC_2}{ddl_1-ddl_2}\) | \(F_{1-2}\) | \(pval_{1-2}\) |
| 3 | Residus | n-\(\Sigma ddl\) |
6.2.3 Interaction entre les facteurs
Examinons les deux modèles suivants. Dans la spécification du modèle, seul l’ordre d’introduction des variables explicatives varie (d’abord espece dans le premier cas, d’abord enchantement dans le second cas):
mylm_o1=lm(perlimpinpin~espece+enchantement,data=broceliande)
anova(mylm_o1, test="chisq")## Analysis of Variance Table
##
## Response: perlimpinpin
## Df Sum Sq Mean Sq F value Pr(>F)
## espece 3 3299 1100 2.5373 0.05547 .
## enchantement 1 31781 31781 73.3279 < 2e-16 ***
## Residuals 859 372294 433
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1mylm_o2=lm(perlimpinpin~enchantement+espece,data=broceliande)
anova(mylm_o2, test="chisq")## Analysis of Variance Table
##
## Response: perlimpinpin
## Df Sum Sq Mean Sq F value Pr(>F)
## enchantement 1 34562 34562 79.7464 <2e-16 ***
## espece 3 517 172 0.3978 0.7546
## Residuals 859 372294 433
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Vous pouvez constater que l’ordre d’introduction des variables explicatives fait varier les résultats, au point que dans un cas l’espèce semble avoir un effet significatif sur la quantité de poudre de perlimpinpin, et non dans l’autre…
D’un point de vue purement mathématique, cette différence est due au fait que notre plan d’expérience est incomplet, c’est-à-dire que le nombre d’observations par groupe diffère (par groupe défini par espece ET enchantement):
broceliande %>%
group_by(enchantement,espece) %>%
summarise(n=n()) ## # A tibble: 8 x 3
## # Groups: enchantement [?]
## enchantement espece n
## <lgl> <fct> <int>
## 1 F chataignier 237
## 2 F chene 235
## 3 F hetre 159
## 4 F sapin 67
## 5 T chataignier 28
## 6 T chene 116
## 7 T hetre 15
## 8 T sapin 7En l’occurrence, parmi les arbres enchantés, on trouve majoritairement des chênes… Donc quand on prend d’abord en compte l’enchantement, on explique déjà en grande partie la différence liée à l’espèce “chêne” et de ce fait l’apport de cette deuxième variable explicative “espece” semble minime…
Ici, le plus sage semble en fait de prendre en compte dans la spécification du modèle l’existence d’une interaction entre enchantement et espece. Cette interaction s’écrit de la manière suivante:
mylm=lm(perlimpinpin~enchantement+espece+enchantement*espece,
data=broceliande)
anova(mylm)## Analysis of Variance Table
##
## Response: perlimpinpin
## Df Sum Sq Mean Sq F value Pr(>F)
## enchantement 1 34562 34562 79.8482 <2e-16 ***
## espece 3 517 172 0.3983 0.7543
## enchantement:espece 3 1773 591 1.3654 0.2520
## Residuals 856 370521 433
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 16.3 Plusieurs variables explicatives, quantitatives et catégorielles: ANCOVA
Intéressons-nous maintenant au jeu de données brocéliande , et plus particulièrement au lien entre la réponse perlimpinpin et la variable espece (catégorielle), d’une part et la variable age (quantitative), d’autre part. On est donc dans le cadre d’une ANCOVA (pour ANalyse de la COVAriance)
6.3.1 Représentation
ggplot(broceliande,aes(x=age,y=perlimpinpin, col=espece))+
geom_point()+
scale_x_log10()+
geom_smooth(method="lm")+
facet_wrap(~espece)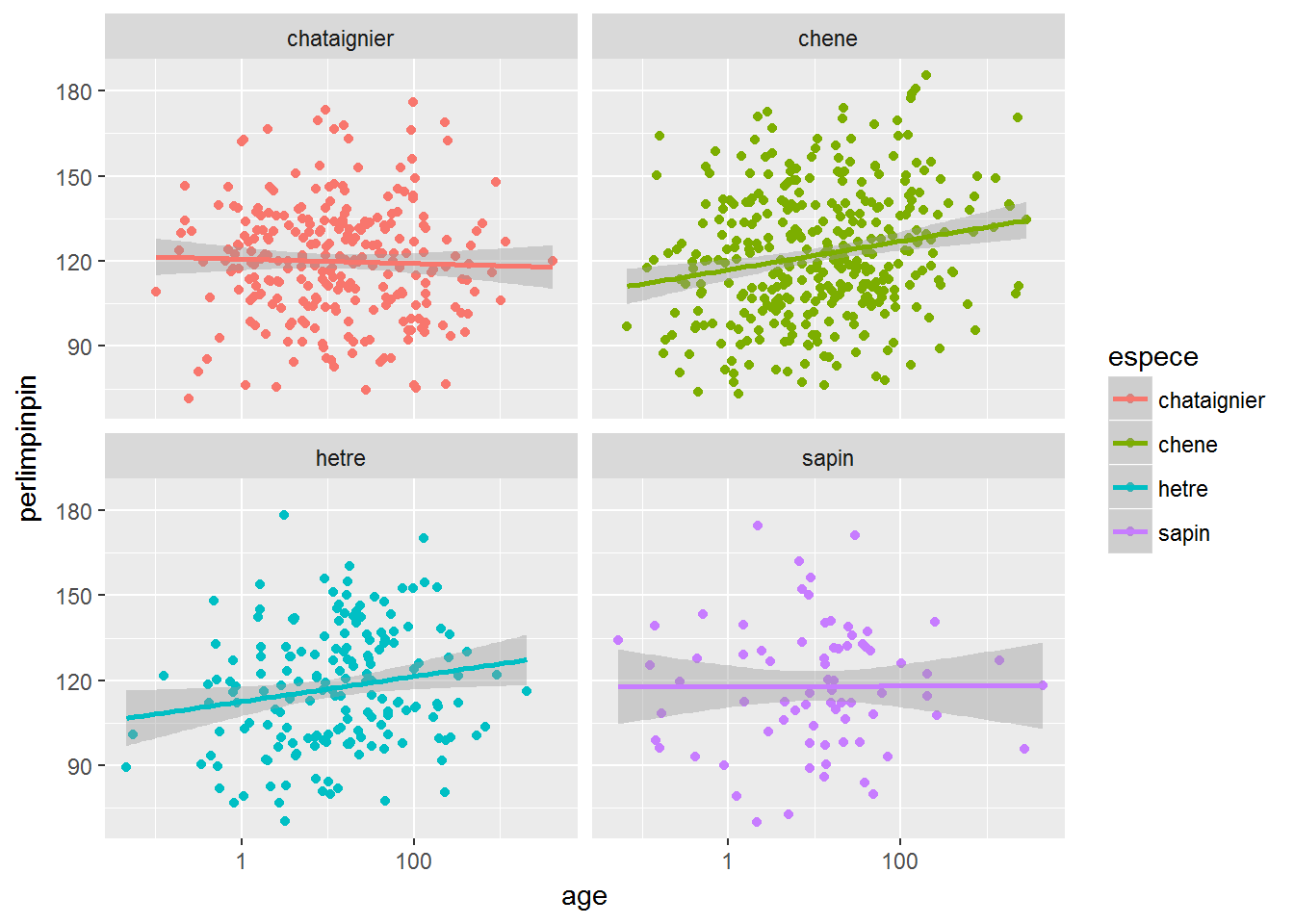
6.3.2 Réalisation de l’ANCOVA
Le graphique renvoyé par les lignes de commande ci-dessus suggèrent que:
especeetagesont liés àperlimpinpinagea un effet surperlimpinpinqui diffère selon le niveau deespece.
Vérifions cette intuition à l’aide d’une ANCOVA:
mylm=lm(perlimpinpin~espece+log(age)+espece*log(age),data=broceliande)
anova(mylm)## Analysis of Variance Table
##
## Response: perlimpinpin
## Df Sum Sq Mean Sq F value Pr(>F)
## espece 3 3299 1099.7 2.3876 0.0676522 .
## log(age) 1 5063 5063.3 10.9936 0.0009527 ***
## espece:log(age) 3 4763 1587.8 3.4475 0.0162814 *
## Residuals 856 394248 460.6
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 16.3.3 Interaction entre les variables explicatives
Comme dans le cas d’une ANOVA à plusieurs facteurs, l’ordre d’introduction des diverses variables explicatives peut modifier le résultat de l’ANCOVA. Consultez la section 6.2.3…